Streptomyces avermitilisのゲノム解析法およびゲノムの特徴
・ゲノム配列決定
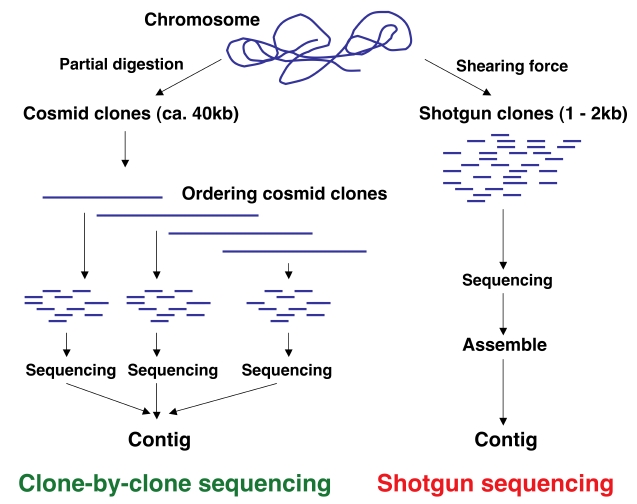
我々がゲノム解析を開始する時点でその解析方法について2つの選択があった。
ひとつは既にS. coelicolor A3(2)で採用された互いにオーバーラップした整列コスミドクローンを一つ一つ決定してい(clone-by-clone)である。
もうひとつはHaemophilus influenzaeのゲノム解析で行った、染色体DNAを1〜2 kbに切断した断片をランダムにシークエンスし、
それらの結果をコンピューターでつなぎあわせる方法“whole genome shotgun sequencingモである。
先のclone-by-clone法は解析当時は一般的ではあったがコスミドクローンの整列化を行わなければならない。
また、Streptomyces属のDNAを含むコスミドクローンは時として大きな欠失を生じやすいことがある。
さらにコスミドクローンの数kbの欠失はゲル電気泳動では確認できず、この欠失を確認することは非常に困難であり、
またこの欠失を知らずにゲノム解析を完成させてしまう危険性がある(実際、S. avermitilisで作製したコスミドクローン
のいくつかで1〜2 kb程度の欠失があることが確認された)。
一方、当時whole genome shotgun sequencing法で決定された最大なゲノムはDeinococcus radiodurans R1であり、
8 Mbを越すゲノムに関しては未知な領域であった。当時進行中であった大腸菌O157堺株(5.6 Mb)がこの方法で行われており、
またclone-by-clone法の危険性を考慮し、S. avermitilisのゲノム解析はwhole genome shotgun sequencing法で行った。
|
| ゲノム解析方法
|
|---|
・ゲノムの特徴
Streptomyces属の菌種は菌糸および胞子ともに耐熱性を示さないがその染色体のGC含量は極めて高く70%以上である。
また、ゲノムサイズも巨大であり、大腸菌や枯草菌のおよそ2倍程度の8Mb以上と見積もられていた。さらに当時、Streptomyces属のいくつか
の菌株で原核細胞生物にもかかわらず真核細胞生物と同じ線状構造の染色体であることが確認された。
また細胞内に存在するプラスミドについても環状構造を有するもののみならず線状構造のものも報告されている。
現在までに報告された線状染色体を有する原核細胞生物はスピロヘータのBorrelia burgdorferiおよび土壌細菌のAgrobacterium tumefaciensである。
これらの細菌のゲノムの末端は両鎖が共有結合したループ構造を形成しているが、一方Streptomyces属のゲノムの末端は
これらの細菌とは異なり両鎖は互いに結合しておらず特異なタンパク質(おそらく5'末端に共有結合している)が結合していることが明らかになった。
これはアデノウイルスや枯草菌φ29ファージのゲノムと同じである。
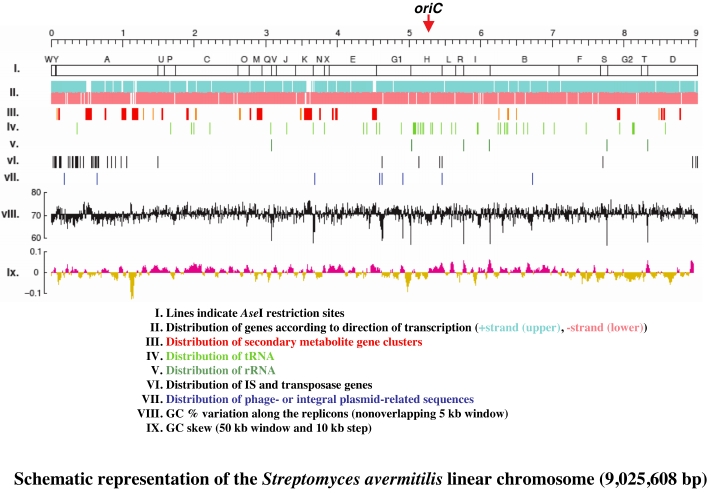
S. avermitilisの染色体は全長9,025,608 bpからなる線状構造を有しており、GC含量は70.7%であった。
本菌には250 kbと94.3 kbのプラスミドが存在しており、両プラスミドともに染色体と同様に線状構造を有している。
S. avermitilisの染色体には7,577個のタンパク質がコードされており、これらは染色体の86.2%を占めている。
その他6つのrRNA (16S-23S-5S)、68個のtRNAおよび1個のtmRNA遺伝子が存在する。
タンパク質をコードする遺伝子の総数は下等真核生物である出芽酵母(6,294個)および分裂酵母(4,824個)のそれらよりも多い。
S. avermitilisの染色体にコードされている遺伝子産物はBLASTおよびpfam解析によって、およそ3分の2(66.1%; 5,014個)
については公開されている他の生物の遺伝子産物の相同性から生物学的な機能の推定が可能であったが、
33.9%(2,563)の遺伝子産物は他の生物の遺伝子産物との相同性は認められるもののhypothetical proteinあるいは
unkonown function proteinとの相同性が示されたため、それらの機能を推定することはできなかった。
S. avermitilis染色体にコードされている遺伝子産物について、互いの相同性を解析した結果、興味あることに7,577個の遺伝子産物のうちの
34% (2,563個)が同一遺伝子から派生したと推定される“パラログ”(重複によりある生物に2つ以上コピーが存在する遺伝子)であることがわかった。
これらのパラログは694に分類され、それぞれのファミリーには2個から89個の遺伝子が含まれている。このようにS. avermitilis
の染色体に存在する遺伝子のおよそ1/3がパラログであることは本菌が進化の過程で高い頻度で染色体上の遺伝子の重複を生じていたことが推察できる。
また、Streptomyces属の染色体は原核細胞生物の中でも最大に属する大きさであり、このようなゲノムの巨大化の一つの要因としてこれら重複遺伝子
の存在があげられるかもしれない。
694種のパラログのうち最も多くの遺伝子が分類されるパラログファミリーは物質の透過に関与するABCトランスポーターをコードする遺伝子群であった。
さらにその次に多くの遺伝子が分類されるパラログファミリーは2成分制御系の遺伝子群であった。その他、制御系に関与するパラログが比較的多く見出された。
多様な生育環境に適応できる緑膿菌でも同様にゲノムの巨大化と制御遺伝子の増加が報告されている。放線菌は代表的な土壌細菌であり、
肥沃な土壌1gには100万個あるいはそれ以上という膨大な数の放線菌が生息している。またこれらの土壌放線菌の大部分はStreptomyces属である。
土壌中のような生息環境は栄養源、温度あるいは湿度など極めて多様性に富む場所である。このような環境に適応して生きていくには、個々の状況に応答し、
それに即した代謝系あるいは透過系を即座に駆動できなければならない。このように多様な環境下に生息する微生物は環境に適応するため、
より多くの制御系遺伝子および物質透過系を保持する必要性から、これらのパラログファミリーに属する遺伝子の重複が進化の過程で行われてきたものかもしれない。
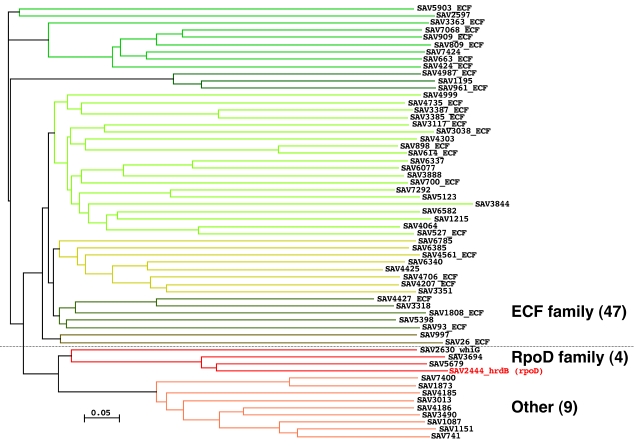
649種に分類されるパラログには極めて興味深い遺伝子の存在がいくつか見出される。大腸菌や枯草菌と同様に放線菌のDNA依存RNAポリメラーゼはα、α、β、β'の
コア酵素複合体からなり、これにσ因子が結合して転写が開始される。S. avermitilisの染色体上には合計64個のσ因子が存在しおり、
これらのほとんどは5つのパラログファミリーに分類された(大部分はECF-σ因子に分類される)。このことはこれらのσ因子がそれぞれのプロモーター配列を認識し、
極めて多様な遺伝子発現制御を可能にしていることが推察される。
|
| S. avermitilisのシグマ因子群の系統解析
|
|---|
・染色体末端
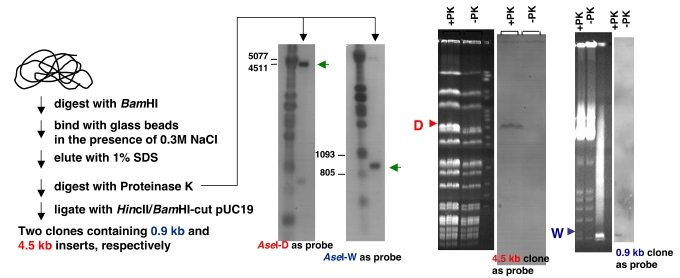
数種のStreptomyces属の菌株から染色体末端部分(テロメア)が上記のタンパク質-DNA複合体の性質を利用した方法によってクローン化されている。
すなわち、タンパク質-DNA複合体が適当なイオン強度のもとでガラスビーズやニトロセルロースに結合する性質を有しており、
このように単離されたタンパク質-DNA複合体をプロテアーゼあるいはピペリジンで処理することによってタンパク質を分解除去し、DNA部分をクローニングすることができる。
|
| 染色体末端部分の検出
|
|---|
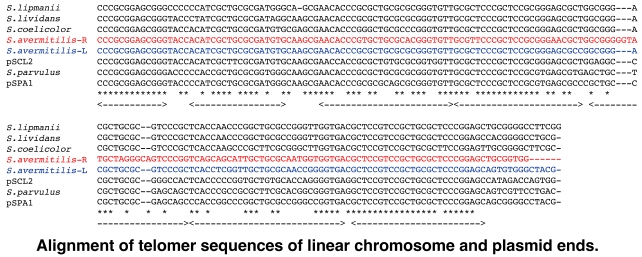
他のStreptomyces属の染色体末端部分(telomer)の塩基配列とS. avermitilisのtelomer部分の塩基配列のアラインメント解析を行ってみると
末端からおよそ90塩基までに極めて相同な配列が認められた。
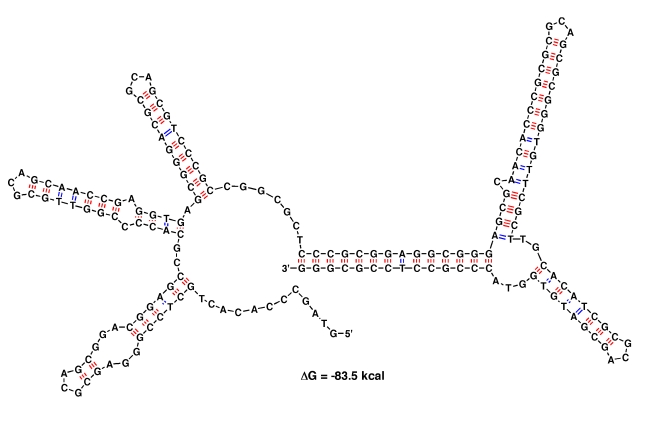
また、これらのtelomer部分は少なくとも7つの逆向き相同配列が存在し、
強固な2次構造を形成するものと推察されている。線状染色体が複製する場合、染色体中央部分から複製が開始されると推察されるが、
合成鎖側の5'末端部分は複製の過程ではDNA合成が行われない。おそらく特殊な末端配列部分の合成によってこの部分の修復が行われるものと考えられるが、
この末端部分の2次構造および5'末端に結合しているDNA-タンパク質複合体が末端部分の修復のためのDNA合成に関与しているかもしれない。
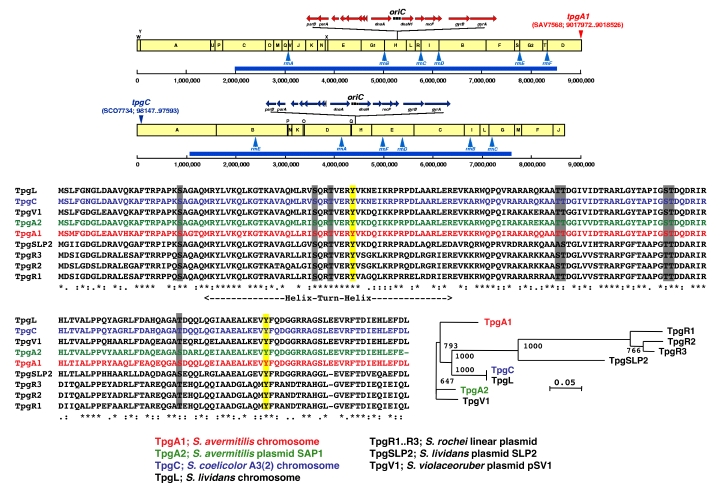
なお、線状ゲノムの末端と結合するタンパク質はCohenらによってS. rocheの線状プラスミドから単離され、さらにその遺伝子も同定された。
この末端結合タンパク質のアミノ酸配列からS. avermitilisおよびS. coelicolor A3(2)のゲノム配列のデータを検索するとアミノ酸配列
80%以上の相同性を有する遺伝子産物がそれぞれのゲノムから一つずつみつかった。これらのタンパク質のアミノ酸配列は高度に保存されており、
N末端側に近い部分にDNA結合ドメインとして知られているhelix-turn-helix構造特有の配列が存在していた。
これらのタンパク質はDNAとおそらくリン酸エステルを介して結合しているものと思われるが、どのSer, ThrあるいはTyrがこの結合に関与しているのかは不明である。